10.5 Preparando los insumos para STAN
- Dividir la base de datos en dominios observados y no observados
Dominios observados.
data_dir <- base_FH %>% filter(!is.na(T_pobreza))Dominios NO observados.
data_syn <-
base_FH %>% anti_join(data_dir %>% select(dam2))
tba(data_syn[,1:8] %>% slice(1:10))| dam2 | Rd | T_pobreza | n_effec | varhat | modificacion_humana | accesibilidad_hospitales | accesibilidad_hosp_caminado |
|---|---|---|---|---|---|---|---|
| 2702 | NA | NA | NA | NA | 0.8018 | -0.7175 | -0.7444 |
| 2901 | NA | NA | NA | NA | 0.0542 | -0.7779 | -0.7341 |
| 1303 | NA | NA | NA | NA | -0.5140 | 0.5302 | 0.4079 |
| 1804 | NA | NA | NA | NA | -0.0336 | -0.6339 | -0.2478 |
| 2404 | NA | NA | NA | NA | 0.3128 | -0.7926 | -0.7880 |
| 2601 | NA | NA | NA | NA | -0.2175 | 1.4552 | 0.9138 |
| 2905 | NA | NA | NA | NA | 0.1913 | -0.3997 | -0.5567 |
| 0202 | NA | NA | NA | NA | -0.3758 | 0.0000 | 0.1482 |
| 0402 | NA | NA | NA | NA | -0.3661 | -0.0114 | -0.2863 |
| 0404 | NA | NA | NA | NA | -0.5678 | 1.0735 | 0.9856 |
- Definir matriz de efectos fijos.
## Dominios observados
Xdat <- cbind(inter = 1,data_dir[,names_cov])
## Dominios no observados
Xs <- cbind(inter = 1,data_syn[,names_cov])- Creando lista de parámetros para
STAN
sample_data <- list(
N1 = nrow(Xdat), # Observados.
N2 = nrow(Xs), # NO Observados.
p = ncol(Xdat), # Número de regresores.
X = as.matrix(Xdat), # Covariables Observados.
Xs = as.matrix(Xs), # Covariables NO Observados
y = as.numeric(data_dir$T_pobreza),
sigma_e = sqrt(data_dir$varhat)
)- Compilando el modelo en
STAN
library(rstan)
fit_FH_arcoseno <- "Recursos/Día3/Sesion3/Data/modelosStan/15FH_arcsin_normal.stan"
options(mc.cores = parallel::detectCores())
model_FH_arcoseno <- stan(
file = fit_FH_arcoseno,
data = sample_data,
verbose = FALSE,
warmup = 500,
iter = 1000,
cores = 4
)
saveRDS(model_FH_arcoseno,
"Recursos/Día3/Sesion3/Data/model_FH_arcoseno.rds")model_FH_arcoseno <- readRDS("Recursos/Día3/Sesion3/Data/model_FH_arcoseno.rds")10.5.1 Resultados del modelo para los dominios observados.
En este código, se cargan las librerías bayesplot, posterior y patchwork, que se utilizan para realizar gráficos y visualizaciones de los resultados del modelo.
A continuación, se utiliza la función as.array() y as_draws_matrix() para extraer las muestras de la distribución posterior del parámetro theta del modelo, y se seleccionan aleatoriamente 100 filas de estas muestras utilizando la función sample(), lo que resulta en la matriz y_pred2.
Finalmente, se utiliza la función ppc_dens_overlay() de bayesplot para graficar una comparación entre la distribución empírica de la variable observada pobreza en los datos (data_dir$pobreza) y las distribuciones predictivas posteriores simuladas para la misma variable (y_pred2). La función ppc_dens_overlay() produce un gráfico de densidad para ambas distribuciones, lo que permite visualizar cómo se comparan.
library(bayesplot)
library(patchwork)
library(posterior)
y_pred_B <- as.array(model_FH_arcoseno, pars = "theta") %>%
as_draws_matrix()
rowsrandom <- sample(nrow(y_pred_B), 100)
y_pred2 <- y_pred_B[rowsrandom, ]
ppc_dens_overlay(y = as.numeric(data_dir$Rd), y_pred2)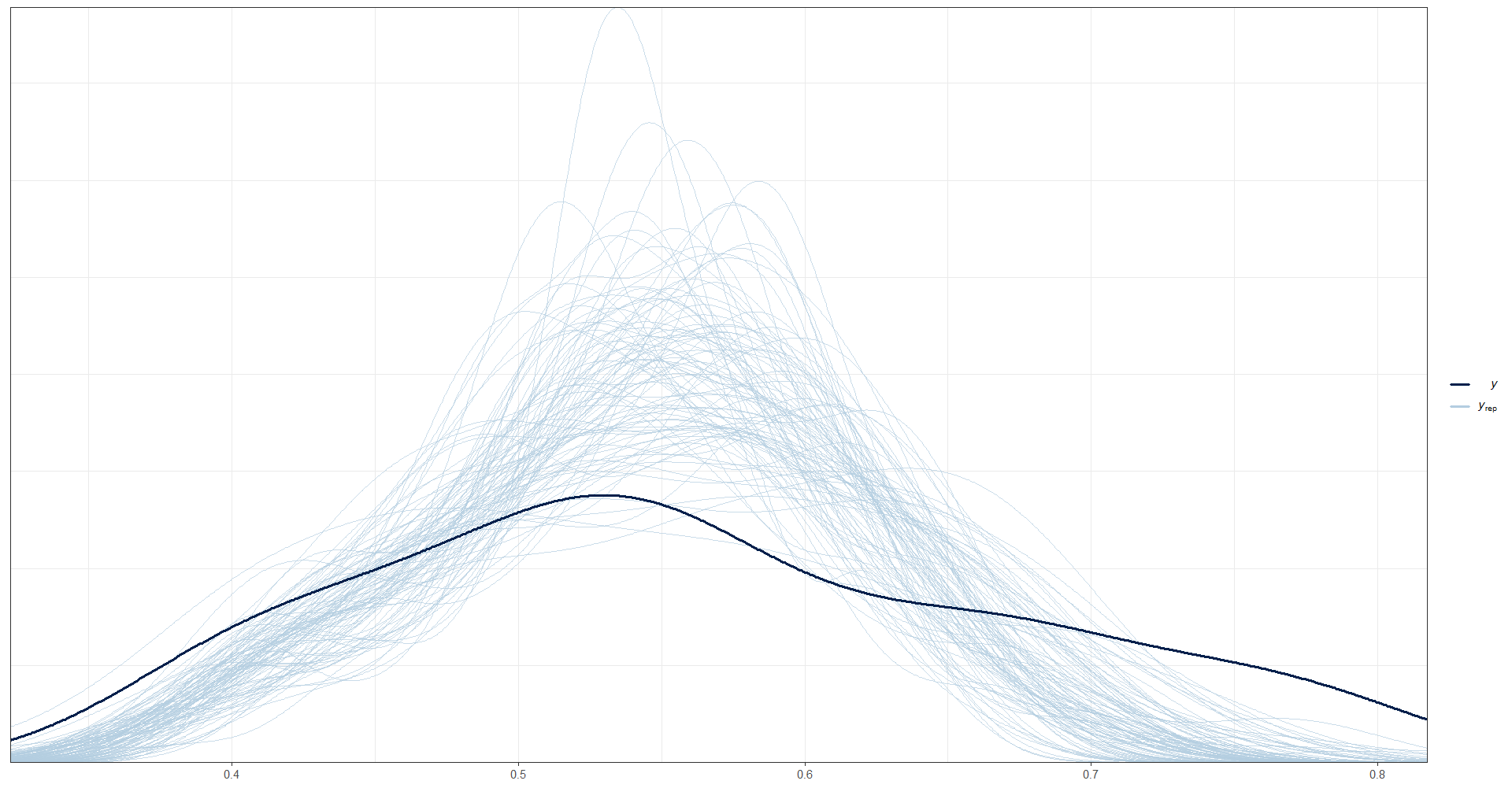
Análisis gráfico de la convergencia de las cadenas de \(\sigma^2_u\).
posterior_sigma2_u <- as.array(model_FH_arcoseno, pars = "sigma2_u")
(mcmc_dens_chains(posterior_sigma2_u) +
mcmc_areas(posterior_sigma2_u) ) /
mcmc_trace(posterior_sigma2_u)
# traceplot(model_FH_arcoseno,pars = "sigma2_u",inc_warmup = TRUE)
# stan_plot(model_FH_arcoseno)
#traceplot(model_FH_arcoseno,pars = "beta",inc_warmup = TRUE)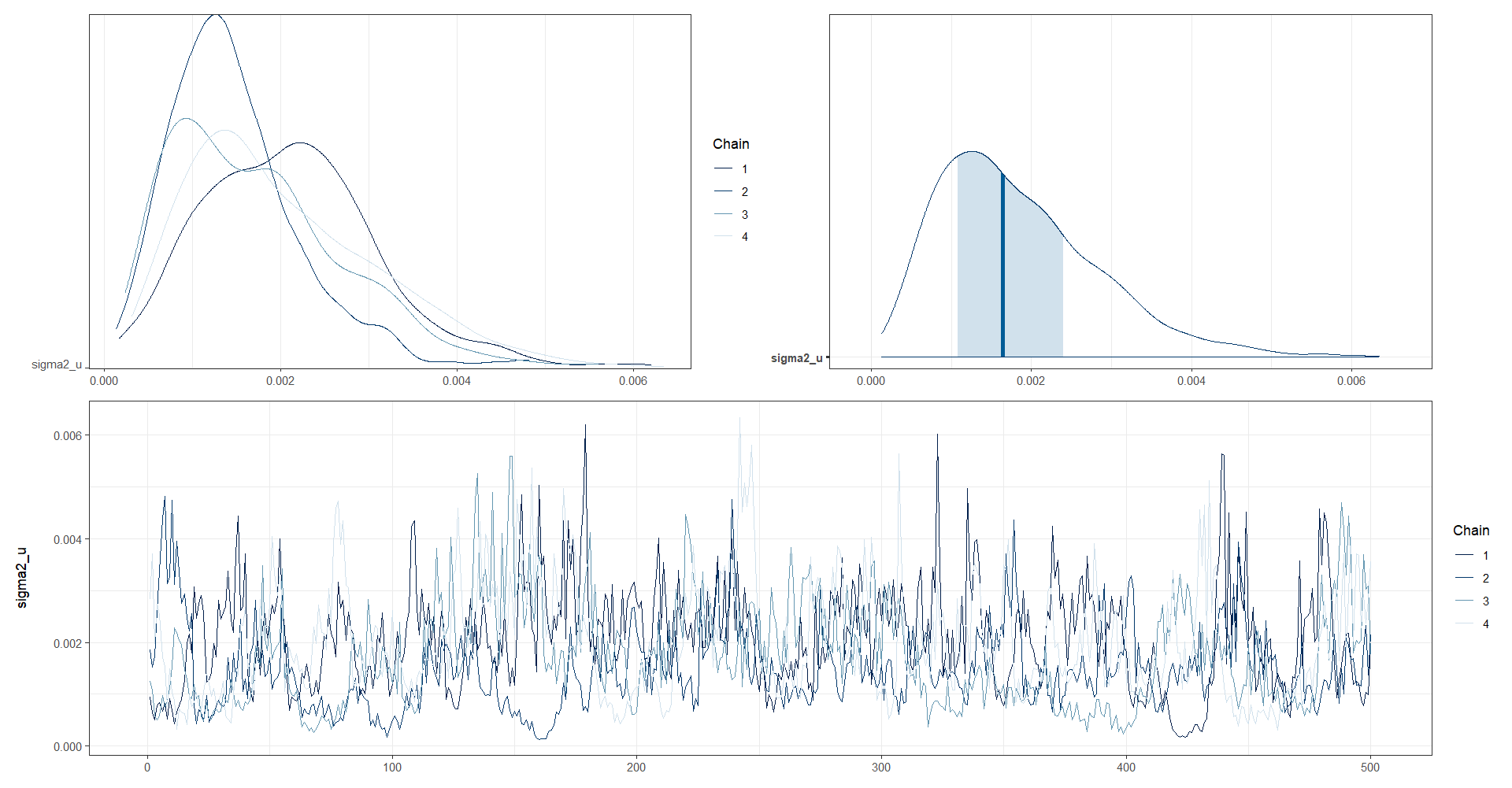
Estimación del FH de la pobreza en los dominios observados.
theta_FH <- summary(model_FH_arcoseno,pars = "theta")$summary %>%
data.frame()
data_dir %<>% mutate(pred_arcoseno = theta_FH$mean,
pred_arcoseno_EE = theta_FH$sd,
Cv_pred = pred_arcoseno_EE/pred_arcoseno)Estimación del FH de la pobreza en los dominios NO observados.
theta_FH_pred <- summary(model_FH_arcoseno,pars = "theta_pred")$summary %>%
data.frame()
data_syn <- data_syn %>%
mutate(pred_arcoseno = theta_FH_pred$mean,
pred_arcoseno_EE = theta_FH_pred$sd,
Cv_pred = pred_arcoseno_EE/pred_arcoseno)